

Stratégies de petites molécules pour la découverte de médicaments ciblés sur l’ARN
Présentation des composés ciblés sur l’ARN
Qu’est-ce que la découverte ciblée de médicaments à ARN ?
La structure 3D complexe et dynamique de l’ARN joue un rôle central dans la biologie humaine et contribue à la pathologie des maladies. Les efforts visant à cibler thérapeutiquement ces structures d’ARN avec de petites molécules, bien que naissants, progressent rapidement. Les principaux progrès comprennent le perfectionnement des outils informatiques permettant de prédire les structures d’ARN conservées et la mise en œuvre de stratégies visant à élargir la portée thérapeutique.
Les interventions actuelles sur les petites molécules utilisent divers mécanismes, notamment la direction de l’épissage (par exemple, Branaplam et Risdiplam approuvés par la FDA), l’inhibition de la traduction de protéines difficiles et la désactivation des structures fonctionnelles dans les ARN non codants.
Services de découverte de médicaments ciblés sur la structure de l’ARN
Il existe différentes méthodes et stratégies pour identifier les liants d’ARN à petites molécules dans le cadre de la découverte de médicaments :
Cibler l'ARN avec de petites molécules
Traditionnellement, les efforts de découverte de médicaments se sont principalement concentrés sur le ciblage des protéines, mais le développement de petites molécules qui interagissent avec les molécules d’ARN suscite un intérêt croissant. Ces molécules peuvent moduler la fonction de l’ARN en modifiant le repliement, la stabilité ou les interactions de l’ARN avec d’autres molécules.
Structure et fonction de l'ARN
Les molécules d’ARN peuvent adopter des structures tridimensionnelles complexes qui jouent des rôles essentiels dans les processus cellulaires tels que l’expression des gènes et la synthèse des protéines. De petites molécules peuvent être conçues pour reconnaître et se lier spécifiquement à ces structures d’ARN, modulant ainsi leur fonction.
Approches de dépistage
Le criblage à haut débit (HTS), le criblage basé sur des fragments et les méthodes informatiques in silico sont couramment utilisés dans la découverte de petites molécules liant l’ARN. HTS consiste à tester de grandes bibliothèques de petites molécules pour identifier les composés qui présentent une affinité et une spécificité de liaison pour l’ARN cible. Les méthodes informatiques, telles que les simulations d’amarrage moléculaire et de dynamique moléculaire, peuvent aider à prédire les modes de liaison et l’affinité des petites molécules avec les cibles d’ARN.
Classes cibles d’ARN
Les molécules d’ARN ciblées pour la découverte de médicaments peuvent inclure l’ARN messager (ARNm), les ARN non codants (tels que les microARN et les ARN longs non codants), l’ARN ribosomal (ARNr), l’ARN de transfert (ARNt) et l’ARN viral. Chaque classe cible d’ARN présente des défis et des opportunités uniques pour la découverte de médicaments à petites molécules.
Applications thérapeutiques
Les petites molécules liant l’ARN ont le potentiel d’être développées en thérapies pour un large éventail de maladies. Par exemple, les petites molécules qui ciblent l’ARN viral peuvent inhiber la réplication virale et sont étudiées comme agents antiviraux. De même, de petites molécules qui modulent la fonction des ARN non codants associés à une maladie sont étudiées comme traitements potentiels du cancer et d’autres troubles.
Traditionnellement, les efforts de découverte de médicaments se sont principalement concentrés sur le ciblage des protéines, mais le développement de petites molécules qui interagissent avec les molécules d’ARN suscite un intérêt croissant. Ces molécules peuvent moduler la fonction de l’ARN en modifiant le repliement, la stabilité ou les interactions de l’ARN avec d’autres molécules.
Les molécules d’ARN peuvent adopter des structures tridimensionnelles complexes qui jouent des rôles essentiels dans les processus cellulaires tels que l’expression des gènes et la synthèse des protéines. De petites molécules peuvent être conçues pour reconnaître et se lier spécifiquement à ces structures d’ARN, modulant ainsi leur fonction.
Le criblage à haut débit (HTS), le criblage basé sur des fragments et les méthodes informatiques in silico sont couramment utilisés dans la découverte de petites molécules liant l’ARN. HTS consiste à tester de grandes bibliothèques de petites molécules pour identifier les composés qui présentent une affinité et une spécificité de liaison pour l’ARN cible. Les méthodes informatiques, telles que les simulations d’amarrage moléculaire et de dynamique moléculaire, peuvent aider à prédire les modes de liaison et l’affinité des petites molécules avec les cibles d’ARN.
Les molécules d’ARN ciblées pour la découverte de médicaments peuvent inclure l’ARN messager (ARNm), les ARN non codants (tels que les microARN et les ARN longs non codants), l’ARN ribosomal (ARNr), l’ARN de transfert (ARNt) et l’ARN viral. Chaque classe cible d’ARN présente des défis et des opportunités uniques pour la découverte de médicaments à petites molécules.
Les petites molécules liant l’ARN ont le potentiel d’être développées en thérapies pour un large éventail de maladies. Par exemple, les petites molécules qui ciblent l’ARN viral peuvent inhiber la réplication virale et sont étudiées comme agents antiviraux. De même, de petites molécules qui modulent la fonction des ARN non codants associés à une maladie sont étudiées comme traitements potentiels du cancer et d’autres troubles.
Oncodesign Services & Zobio : une alliance solide de partenaires pour fournir des services expérimentés pour le développement ciblé sur l’ARN
Oncodesign Services est une CRO leader spécialisée dans le développement de médicaments et les services précliniques. En 2024, Oncodesign Services a réalisé l’acquisition de Zobio, une plateforme leader dédiée à la découverte de petites molécules. Les 2 partenaires proposent une offre totalement intégrée pour accompagner les programmes innovants depuis la validation des cibles jusqu’à la sélection des candidats.
Criblage phénotypique dans la découverte de médicaments à petites molécules ciblant l’ARN
Dans le contexte de la découverte de liants d’ARN à petites molécules, le criblage phénotypique est une approche précieuse qui consiste à évaluer les effets de petites molécules sur des phénotypes cellulaires ou organismes sans connaissance préalable de la cible.
- Large potentiel de découverte : les analyses phénotypiques offrent un moyen d’identifier les petites molécules qui modulent la fonction de l’ARN sans hypothèses préalables sur la cible spécifique ou le mécanisme d’action. Cette approche peut découvrir des liens inattendus entre les petites molécules et les cibles d’ARN, conduisant ainsi à la découverte de nouveaux candidats thérapeutiques.
- Capturer les interactions complexes : les molécules d’ARN interagissent souvent avec plusieurs protéines et composants cellulaires pour exercer leurs fonctions biologiques.
Les cellules choisies peuvent être conçues, si nécessaire, soit pour imiter l’état de « maladie » souhaité, soit pour permettre un criblage à haut débit.
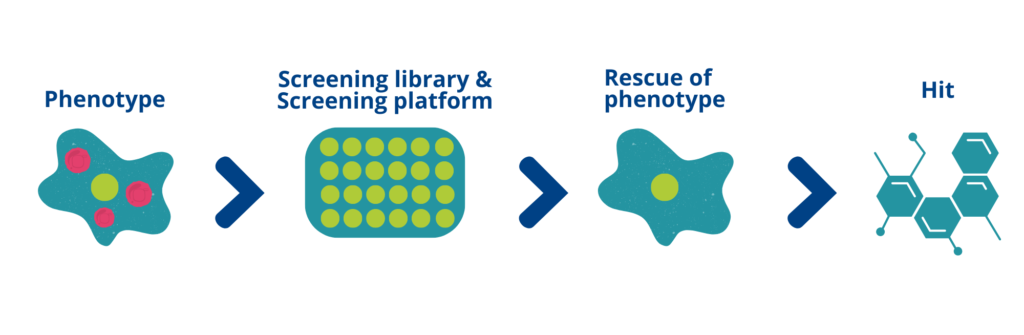
Développer et réaliser des criblages phénotypiques à travers diverses méthodes
Approche indépendante de la cible : contrairement aux criblages basés sur des cibles, les criblages phénotypiques ne nécessitent pas de connaissance préalable de la cible ARN spécifique impliquée dans la pathologie de la maladie. Ceci est particulièrement avantageux lors de l’étude de réseaux complexes de régulation de l’ARN ou lorsque la cible est inconnue ou mal caractérisée.
Example of a target agnostic approach
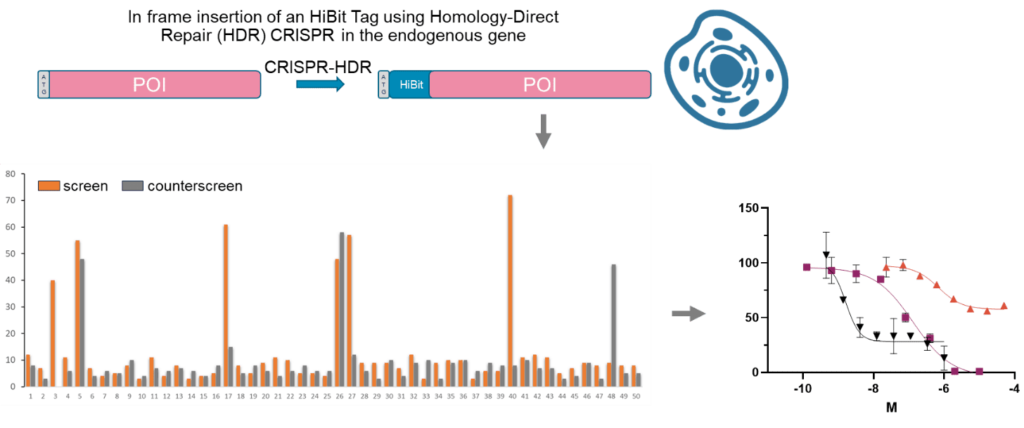
Criblage phénotypique basé sur des cibles : les lignées cellulaires peuvent être manipulées génétiquement avec précision pour s’aligner sur un phénotype souhaité. Ce processus de génie génétique permet le développement de lignées cellulaires sur mesure pour les protocoles de criblage à haut débit (HTS), facilitant ainsi des stratégies de criblage efficaces et précises pour identifier des composés ayant des effets thérapeutiques spécifiques.
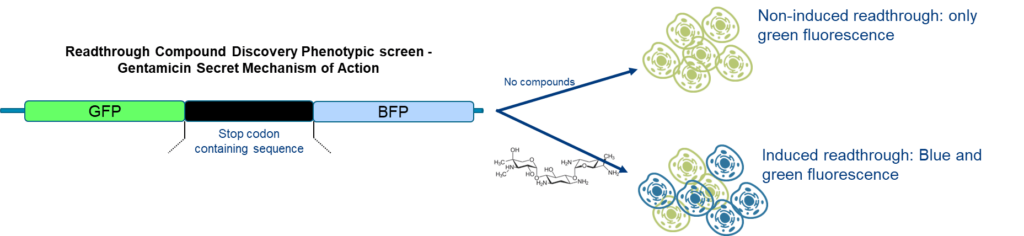
Fournir des méthodes biophysiques pour découvrir ou caractériser les liants d’ARN
Les méthodes biophysiques jouent un rôle unique dans la découverte de médicaments basée sur des cibles, permettant une caractérisation cinétique, thermodynamique et structurelle détaillée des interactions cibles de petites molécules. Ces méthodes sont généralement sans marquage et détectent une interaction directe entre de petites molécules et un ARN cible, convenant à la fois comme approches de criblage primaire et comme tests de validation.
- Résonance Plasmon de Surface (SPR)
La SPR est une méthode directe et à débit modéré pour détecter la liaison de petites molécules à l’ARN. Dans SPR, un partenaire de liaison (dans ce cas, l’ARN cible) est immobilisé sur une surface en or fonctionnalisée et l’autre partenaire de liaison circule sur la surface en solution. L’interaction de liaison entraîne un changement d’indice de réfraction sur la surface qui est détecté comme une réponse SPR. Utilisant la microfluidique à flux continu, la méthode surveille en temps réel l’interaction des petites molécules injectées sur la cible immobilisée. Utilisant une quantité relativement faible d’ARN, un instrument SPR moderne à haut débit permet d’analyser jusqu’à 1 000 interactions par jour. Les résultats identifiés à partir du criblage primaire sont en outre confirmés par titrage dans l’ARN cible immobilisé pour la détermination de l’affinité. En plus d’être sans étiquette, le principal avantage du SPR est sa capacité à mesurer à la fois l’affinité (KD) et la cinétique (ka et kd) de liaison en temps réel (Figure 1). L’affinité et les paramètres cinétiques sont des informations précieuses qui permettent aux chimistes médicinaux de concevoir des composés optimisés dotés de propriétés médicamenteuses améliorées.
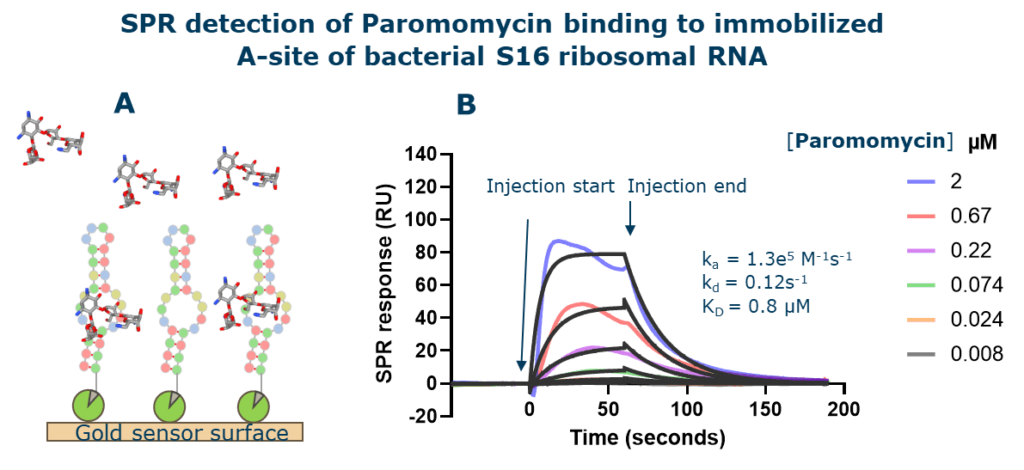
Figure 1: Surveillance de l’interaction de la paromomycine se liant à un ARN ribosomal bactérien S16 immobilisé. A) L’hélice 44 biotinylée de l’ARN ribosomal bactérien S16 a été immobilisée sur une puce sensorielle en or recouverte de streptavidine. Une concentration croissante de paromomycine a été injectée sur la surface. B) L’interaction de la paromomycine avec l’ARNr a entraîné une réponse SPR qui a été suivie dans le temps (traces colorées). Les traces SPR résultantes sont appelées sensorgrammes et peuvent être adaptées à un modèle cinétique (traces noires) pour déterminer l’affinité et la cinétique de liaison.
- Spectroscopie RMN
La spectroscopie RMN est une technique basée sur des solutions à faible débit, mais puissante, qui peut être utilisée pour surveiller les interactions ARN-petites molécules (à la fois faibles et étroites), pour cartographier les sites de liaison, pour évaluer le repliement de l’ARN et pour déterminer les structures à haute résolution. et caractériser la dynamique conformationnelle.
Les interactions ARN – petites molécules sont facilement identifiées dans des spectres 1D 1H simples qui peuvent se concentrer soit sur les signaux du ligand (RMN observée par le ligand), soit sur les signaux de l’ARN (RMN observée par l’ARN). Ce dernier fournira également des informations à basse résolution sur le repliement et l’intégrité de la molécule d’ARN (Figure 2).
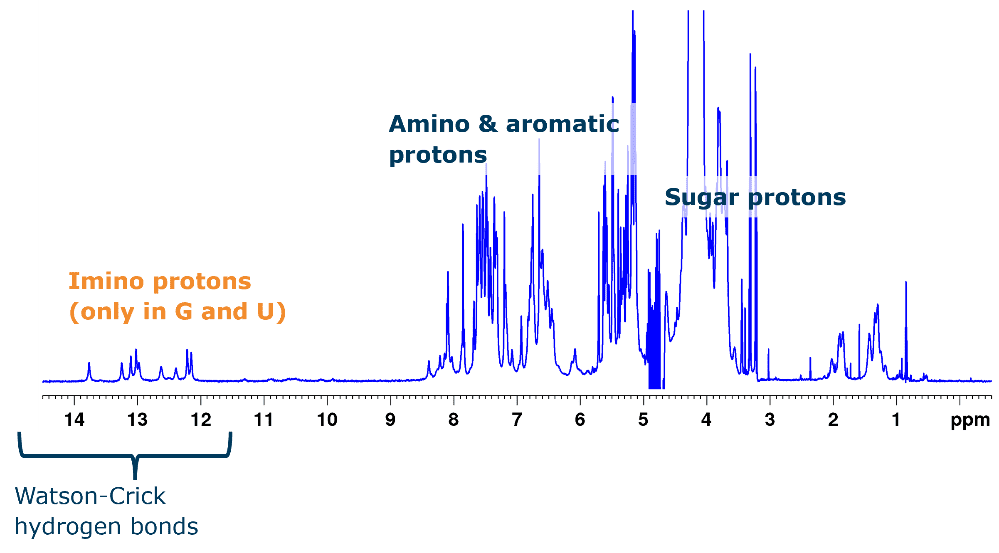
Figure 2: 1D 1H spectrum of HIV-1TAR stem loop (29 nt). Typical 1H chemical shifts for different groups are marked. The distinct chemical shift of imino protons involved in Watson-Crick base pairs provides information on folding.
Grâce aux expériences de RMN 2D et 3D et au marquage avec des isotopes stables (13C et 15N), les résonances (pics) des spectres d’ARN peuvent être attribuées, ouvrant la voie à des analyses de plus en plus détaillées : le site de liaison de petites molécules d’intérêt peut être cartographié, le l’effet des ligands sur les conformations locales peut être évalué et les structures à haute résolution des complexes peuvent être déterminées (Figure 3).
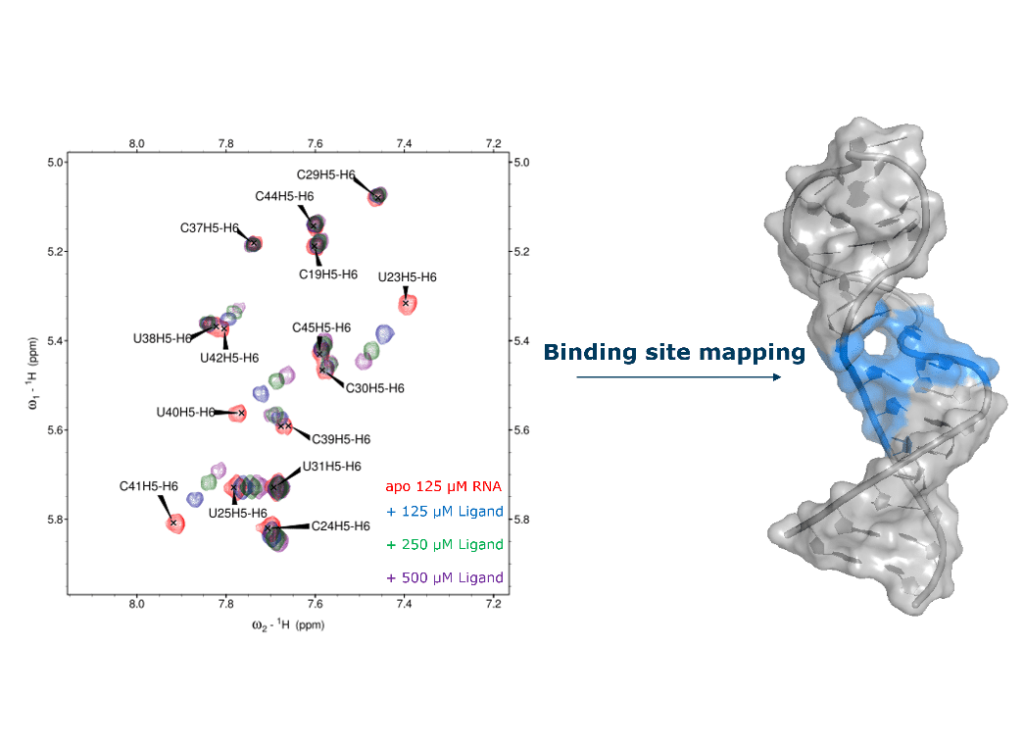
Figure 3: Binding site mapping of acetylpromazine. (left) Titration of HIV-1 TAR stem loop (29 nt) with acetylpromazine : 2D 1H, 1H TOCSY spectra of HIV-1 TAR in the presence of increasing amounts of acetylpromazine. (right) The bases that show chemical shift perturbations upon addition of ligand are highlighted in blue on the structure of HIV-1 TAR, delineating the binding site for acetylpromazine.